一个基于深度学习的框架,可用于有效生成细胞内代谢的动力学模型 - IT思维
公众号/ ScienceAI(ID:Philosophyai)
编辑 | 萝卜皮
代谢动力学模型通过机械关系将代谢通量、代谢物浓度和酶水平联系起来,使其对于理解、预测和优化生物体的行为至关重要。然而,由于缺乏动力学数据,传统的动力学建模通常只产生很少或没有具有理想动力学特性的动力学模型,使得分析不可靠且计算效率低下。
瑞士洛桑联邦理工学院(École Polytechnique Fédérale de Lausanne,EPFL)的研究人员提出了 REKINDLE(Reconstruction of Kinetic Models using Deep Learning),这是一个基于深度学习的框架,用于有效生成具有与细胞中观察到的动态特性相匹配的动力学模型。
研究人员展示了 REKINDLE 使用少量数据在新陈代谢的生理状态中导航的能力,而计算要求显著降低。结果表明,数据驱动的神经网络吸收了代谢网络的隐含动力学知识和结构,并生成了具有定制属性和统计多样性的动力学模型。研究人员表示,该框架可能会促进学界加深对新陈代谢的理解,从而加速未来在生物技术和健康方面的研究。
该研究「Reconstructing Kinetic Models for Dynamical Studies of Metabolism using Generative Adversarial Networks」为题,于 2022 年 8 月 30 日发布在《Nature Machine Intelligence》。

高通量测量技术的技术进步推动了生物技术和医学的发现,使研究人员能够将不同的数据类型整合到细胞状态的表示中,并获得对细胞生理学的见解。从历史上看,研究人员使用基因组规模模型(细胞代谢的数学描述)将实验观察到的数据与细胞表型相关联。
然而,传统的基因组规模模型无法预测细胞对内部或外部刺激的动态反应,因为它们缺乏有关代谢调节和酶动力学的信息。最近,学界已将重点转移到开发动力学代谢模型上,以促进人们对细胞生理学的理解。
动力学模型捕获细胞状态的时间依赖性行为,与通过稳态方法(如通量平衡分析)获得的信息相比,提供有关细胞代谢的额外信息。然而,难以获得(1)每个反应的确切机制和(2)所述机制的参数,例如米氏常数或最大速度,阻碍了动力学模型的建立。
在大多数动力学建模方法中,未知的反应机制是通过近似反应机制来假设或建模的。获得未知参数的主要挑战是生物系统固有的不确定性。由于描述生物系统的数学方程固有的不确定性,该模型通常可以重现多个而不是一组独特的参数值的实验测量值。为了应对这些挑战,研究人员经常采用基于蒙特卡罗抽样的框架。
在这些方法中,EPFL 的研究人员首先通过整合实验测量并确保与物理化学定律的一致性来减少允许参数值的空间。然后对缩减的解空间进行采样以提取替代参数集。
然而,基于采样的动力学建模框架经常会产生与实验观察到的生理学不一致的大量动力学模型亚群。例如,与实验数据相比,构建的模型可能局部不稳定或显示代谢状态的时间演化过快或过慢(图 1)。这导致计算效率的相当大的损失,特别是对于具有理想属性的亚群的低发生率。例如,局部稳定的大规模动力学模型的生成率可能低于 1%。要求其他模型属性,例如实验观察到的代谢状态的时间演变,进一步降低了所需模型的发生率。
事实上,只有一小部分参数空间同时满足所有理想的模型属性,通过观察表明,这个子空间不是连续的。此外,这些方法都不能保证通常作为无偏采样实现的采样过程将产生理想的参数集。这些缺点随着动力学模型大小的增加而被放大,并且在参数空间中找到满足所需特性和观察到的生理学的区域变得具有挑战性。此外,这些区域的结构非常复杂,以至于需要非线性函数逼近器(例如神经网络)来映射它们。


图示:REKINDLE 框架和应用程序概述。(来源:论文)
因此,EPFL 的研究人员提出 REKINDLE(Reconstruction of Kinetic Models using Deep Learning)来应对这些挑战。这种无监督的基于深度学习的方法利用生成对抗网络 (GAN) 来生成动力学模型,以捕捉实验观察到的代谢反应。
REKINDLE 利用现有的动力学建模框架来创建训练 GAN 所需的数据。使用这些神经网络有效地生成具有所需特性的模型大大减少了对传统动力学建模方法所需的大量计算资源的需求。例如,REKINDLE 可用于在几秒钟内在常用硬件上创建大型合成数据集。
重要的是,该团队展示了 REKINDLE 在低数据状态下使用迁移学习导航新陈代谢生理状态的能力,证明了针对一种生理学训练的神经网络可以使用少量数据针对另一种生理学进行微调。REKINDLE 与创建动力学模型的传统方式不同,为更全面的计算研究和高级新陈代谢统计分析铺平了道路。
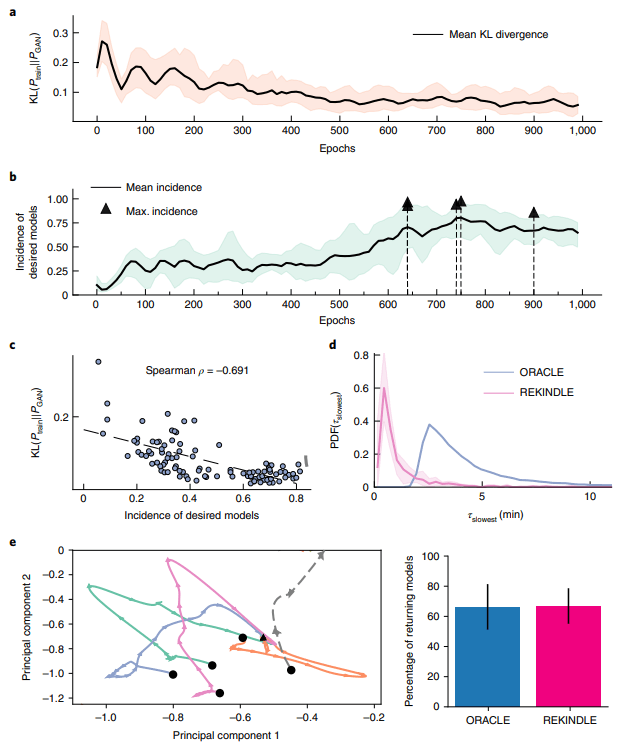
图示:生成和验证 GAN 生成的动力学模型。(来源:论文)
REKINDLE 用于生成生物学相关的动力学模型
REKINDLE 框架由四个连续的步骤组成。
REKINDLE 的输入是从传统动力学建模方法(例如,通过蒙特卡罗采样)获得的动力学参数集。该过程首先测试动力学参数集的生物学相关性。如果从具有该参数集的动力学模型获得的代谢响应具有实验观察到的动态响应,研究人员认为该动力学参数集具有生物学相关性。
然后,将参数集分为生物学相关或不相关的两类(例如,提供过慢、过快或不稳定动态的代谢反应的集合),并相应地标记它们。虽然在这里使用 REKINDLE 生成具有生物学相关动力学的动力学模型,但该框架允许施加其他生化特性或特性和生理条件的组合来构建和标记数据集。然后使用标记的数据集来训练条件 GANs。
条件 GANs 由两个前馈神经网络、生成器和鉴别器组成,它们在训练期间以类标签为条件。训练过程的目标是获得一个良好的生成器,该生成器从特定的预定义类生成动力学模型,这些模型与训练数据中同一类的动力学模型无法区分。
训练完成后,研究人员通过一系列测试验证生成的动力学模型的生物学相关性。首先通过比较它们在参数空间中的分布来测试生成数据和训练数据的统计相似性。然后,检查雅可比(方法)的特征值的分布及其相应的主要时间常数,以验证生成的参数集是否满足所需的动态响应。最后,测试模型对稳态代谢曲线扰动的动态响应,以评估生成的参数集的稳健性。
图示:REKINDLE 生成的集合的可解释性。(来源:论文)
讨论与结论
关于细胞内代谢通量、代谢物浓度和酶的动力学特性的实验验证信息的稀缺性导致具有多个能够捕获实验数据的模型的系统不确定。由于需要大量计算资源来量化所涉及的不确定性,研究人员通常最终只使用众多替代解决方案中的一种,从而导致对细胞代谢行为的不可靠分析和错误预测。这是在代谢研究中有限使用动力学模型的原因之一,尽管它们的能力得到广泛认可。
REKINDLE 提供了一种对参数空间进行采样和创建动力学模型的高效方法,从而为分析这些网络提供了前所未有的全面性,并提供了更广泛的动力学模型适用范围。一般来说,非线性参数空间的采样已成为解决计算物理、生物学和化学中不确定性的标准方法。
研究人员通过概念验证应用程序证明了 REKINDLE 能够学习代谢网络的机械结构,并对与相关模型属性相对应的动力学参数子空间进行分层。通过学习动力学参数的复杂高维空间和相关模型属性之间的映射,GANs 增强了 (1) 根据该团队指定的标准创建模型的效率和 (2) 根据他们的标准划分参数空间的信息。因此,REKINDLE 在生成模型方面比传统方法快几个数量级。
对于从头开始训练的 GANs,REKINDLE 需要约 1,000 个数据点才能可靠地达到相关模型的高发生率,对应于约 15-20 分钟的训练时间。经过训练的 REKINDLE 生成器在约 18 秒内生成 100 万个模型。相比之下,目前最有效的动力学建模框架之一的 ORACLE 在相同的硬件上在 18-24 小时内完成了相同的任务。由于训练数据较少,当通过迁移学习生成模型时,生成时间的减少更为明显。
一旦通过迁移学习为目标生理学训练了生成器,它就可以用于使用新生成的合成数据集扩展传统的小型数据集。这种扩展的数据集适用于传统的统计分析,以进一步了解所研究的系统。与生成动力学模型的传统方法相比,这为 REKINDLE 在应用范围和综合性方面提供了关键优势。

图示:通过迁移学习外推到多种生理学。(来源:论文)
REKINDLE 将允许构建高度策划的「现成」网络库,这些网络已经使用来自标准动力学代谢模型的数据集进行了预训练。这样的存储库将使研究人员能够将该框架应用于从生物技术到医学的不同生理学和研究类型和应用。
总之,研究人员提出了一个框架来利用深度学习的力量生成动力学模型,同时保留传统方法的便利性,研究人员可以在其中分析代谢网络内的结构依赖性、相关性和反馈。REKINDLE 的开放存取代码将允许广大实验人员和建模人员将此框架与实验方法相结合,并受益于所研究生物体的分析和代谢干预的协同方法。
论文链接:https://www.nature.com/articles/s42256-022-00519-y