纳米生物:新的机器学习模型预测无机纳米颗粒如何与蛋白质相互作用 - IT思维
公众号/ ScienceAI(ID)
编辑 | 萝卜皮
随着抗生素耐药性感染的增加以及不断演变扩大的大流行病毒,研究人员希望能够设计出可以关闭这些感染的工程纳米粒子。
已知仿生纳米颗粒可用作纳米级佐剂、酶模拟物和淀粉样蛋白原纤维化抑制剂。它们的进一步发展需要更好地了解它们与蛋白质的相互作用。
关于蛋白质-蛋白质相互作用的丰富知识,可以作为设计蛋白质-纳米粒子组件的指南,但是用于蛋白质-蛋白质相互作用的计算包中的化学和生物输入,不适用于无机纳米粒子。
分析蛋白质复合物的化学、几何和图论描述符,密歇根大学的研究人员发现几何和图论描述符统一适用于生物和无机纳米结构,并且可以预测蛋白质对中的相互作用位点,准确度 >80%,分类概率约为 90%。
研究人员把针对蛋白质-蛋白质相互作用训练的机器学习算法,扩展到无机纳米粒子,并发现实验和预测的蛋白质相互作用位点之间几乎完全匹配。同样,这些发现可以扩展到其他有机和无机纳米粒子,以预测它们与生物分子和其他化学结构的组装,形成锁钥复合物。
该研究以「Unifying structural descriptors for biological and bioinspired nanoscale complexes」为题,于 2022 年 4 月 28 日发布在《Nature Computational Science》。

蛋白质之间的相互作用在概念上被描述为锁钥复合物,反映在多种成功的蛋白质-蛋白质相互作用(PPI)算法中,例如 PRISM、PSIVER 和 MaSIF。这些和其他计算包通过评估潜在「关键」与许多其他「关键」的成对相似性来预测蛋白质复合物的形成和相互作用位点。类似的概念可以应用于纳米粒子 (NP)-蛋白质相互作用,但其实现需要与蛋白质数据库(PDB)相当的庞大的 NP-蛋白质对 X 射线衍射数据库,目前尚不可用。
其他 PPI 算法,例如 SPIDER 和 Pre-PPI,将对接分子的几何描述与生物体水平的结构关系结合起来,例如进化同源性和基因组学中的蛋白质网络。重要的是,这些 PPI 软件包还假设相互作用的分子是来自氨基酸(AA)的线性聚合物。
这样的描述符对于蛋白质来说是自然的,但无法将这些算法扩展到仿生无机纳米粒子,即使它们可能携带一些 AA 作为表面配体。简化的分子输入线输入系统可以注释非肽生物分子的结构,但同样不适用于仿生纳米粒子,即使是那些基于碳原子的纳米粒子,而许多表现出强特定生物活性的纳米粒子完全是无机的。
在原子分子动力学(MD)水平上统一蛋白质和 NP 的结构描述是可能的,这代表了预测 NP-蛋白质相互作用的最新技术。然而,典型的原子 MD方法探测的相互作用时间主要限于数百纳秒。即使使用专用的 Anton2 超级计算机,交互时间也只能达到 2 μs,而蛋白质-蛋白质和 NP-蛋白质复合物形成所需的时间可能超过几分钟甚至几小时。
虽然对于大分子之间的复合物很重要,但以偶极-偶极力和集体氢键为例的弱多中心相互作用,在没有严格的时间限制的情况下难以实施。纳米级相互作用的能量景观的复杂性也可能导致 MD 模拟在形成完全平衡的复合物之前陷入亚稳态。
在这项研究中,研究人员分析了有助于形成蛋白质-蛋白质复合物的不同结构特征的作用,目的是确定可以统一适用于蛋白质和 NPs 之间复合物的结构描述符。识别这些描述符将使人们能够将从大量 PPI 数据集和现有算法中获得的知识扩展到在各种生物医学环境中遇到的 NP-蛋白质对,从药物输送到 NP 的环境影响。

图示:蛋白质复合物距离矩阵的概念和描述符的引入。(来源:论文)
「我们将纳米粒子重新设想为不仅仅是药物输送工具。我们认为它们本身就是活性药物。」该研究的作者之一 J. Scott VanEpps 说。
「通过将数学方法应用于蛋白质-蛋白质相互作用,我们简化了模拟这些对中一种蛋白质的纳米粒子的设计。」该研究的通讯作者 Nicholas Kotov 说。
「纳米颗粒比生物分子更稳定,可以产生全新类别的抗菌和抗病毒药物。」
尽管纳米级结构之间的分子间相互作用很复杂,但 GE + GT 描述符充分预测了蛋白质的复合物和相互作用位点的形成。相同的描述符可以直接应用于 NP。在蛋白质-蛋白质复合物上训练的 ML 算法准确地预测蛋白质-NP 复合物的结构这一事实提供了直接且无可争议的证据,证明了已知具有多种生物功能的水溶性无机 NP 的仿生性质。

图示:构建基于距离的特征矩阵以预测蛋白质复合物。(来源:论文)
纳米碳和其他无机纳米粒子的化学性质可能与蛋白质的化学性质大不相同。虽然它们与蛋白质的复合物的动力学往往难以建模,但开发的 ML 算法可以简化它们的分子设计,用于特定的生物医学或生物制造应用。纳米级物种的刚性水平可以通过 GNM 参数来描述,可以快速准确地计算。
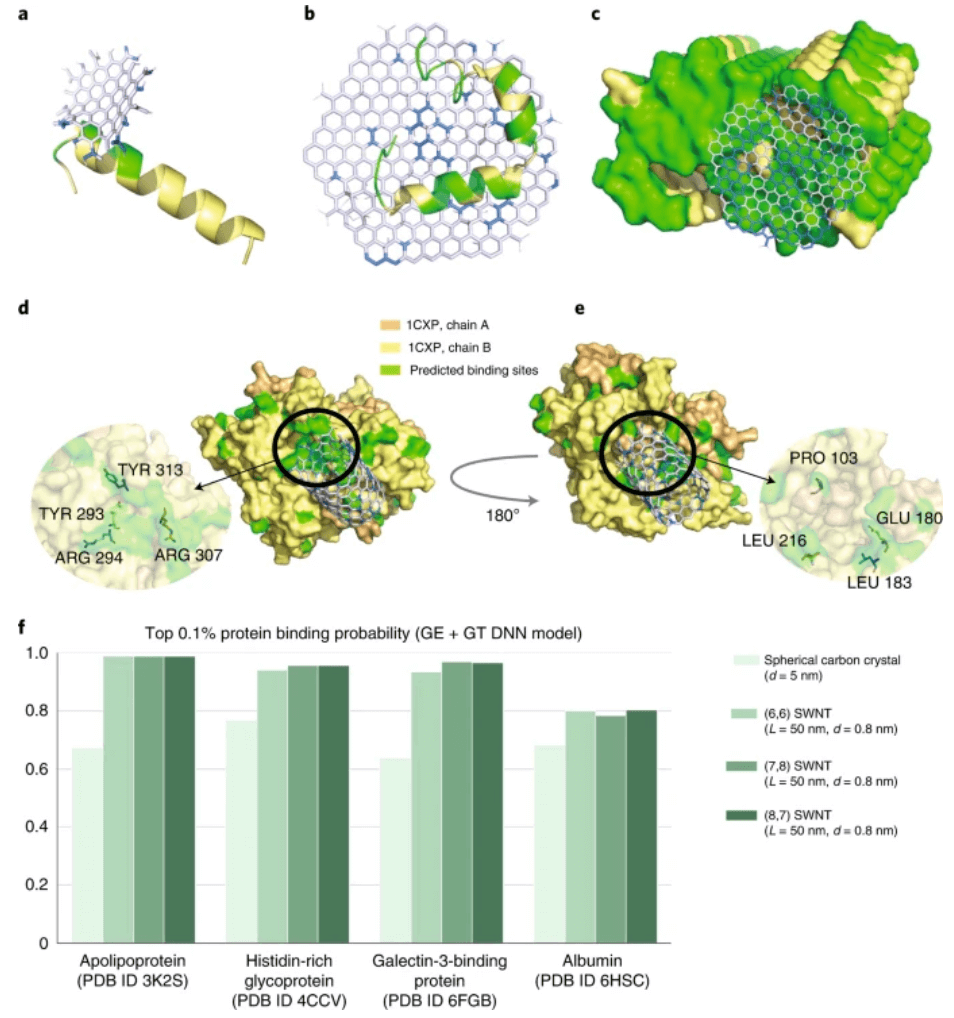
图示:蛋白质和纳米碳复合物的预测。(来源:论文)
GNM 模式的分析有助于 (1) 在不同纳米级物种的光谱中设计分子刚性和 (2) 预测不同温度下的相互作用位点。这两项任务都可以通过使用玻尔兹曼分布,为各种化学键添加基于物理的热运动描述来完成,这将为生物和非生物纳米级物种提供互补的描述。
从基本的角度来看,这些发现扩展了对在纳米级实体之间形成锁键界面的结构要求的理解边界,并整合了拓扑、黎曼几何和多重分形的概念,以建立它们之间的共性。
从实践的角度来看,这些发现为快速设计具有特定形状和表面化学性质的生物纳米结构提供了一个工具箱,用于生物医学和其他应用。
虽然传统的 CH 描述符对蛋白质-蛋白质和蛋白质-NP复合物预测的准确性提供了有限的输入,但研究人员期望随后开发统一的 CH 描述符,包括蛋白质和 NP 之间的非加性和集体效应,例如,局部计算的MD或密度泛函理论计算也将提高此类相互作用位点和亲和常数预测的准确性。
论文链接:https://www.nature.com/articles/s43588-022-00229-w
相关报道:https://phys.org/news/2022-05-nanobiotics-machine-nanoparticles-interact-proteins.html