可在细胞环境中监督挖掘分子模式的卷积网络 - IT思维
公众号/ ScienceAI(ID:Philosophyai)
编辑 | 萝卜皮
低温电子断层扫描可捕获有关细胞和组织分子成分的大量结构信息。对于未经训练的人来说,冷冻电子断层照片看起来更像是沙子中的痕迹,而不是细胞的详细快照。虽然训练有素的专家可以很好地识别和标记断层图中的不同细胞结构,但一代一代培养这样的专家非常耗时。
欧洲分子生物学实验室(European Molecular Biology Laboratory,EMBL)的研究人员开发了 DeepPiCt(deep picker in context),这是一种开源 深度学习 框架,用于低温电子断层扫描中的监督分割和大分子复合物定位。
为了根据实验数据训练和基准化 DeePiCt,该团队全面注释了 20 张粟酒裂殖酵母的断层照片,包括核糖体、脂肪酸合成酶、细胞膜、核孔复合物、细胞器和胞质溶胶。通过将 DeePiCt 与该 数据 集上的最先进方法进行比较,展示了其识别低丰度和低密度复合物的独特能力。
研究人员使用 DeePiCt 来研究细胞核糖体的组成不同的亚群,重点是它们与线粒体和内质网的背景关联。该团队将预先训练的网络应用于 HeLa 细胞断层扫描表明,DeePiCt 在几分钟内就可以在来自不同生物物种的看不见的数据集中实现高质量的预测。
该研究以「Convolutional networks for supervised mining of molecular patterns within cellular context」为题,于 2023 年 1 月 23 日发布在《Nature Methods》。

低温电子断层扫描(cryo-ET)以分子级分辨率生成细胞景观的三维(3D)快照,使得研究大分子复合物在其天然环境中的结构和功能状态成为可能,并揭示不同的大分子群体如何与细胞结构相互作用。通过改进仪器、样品制备方案和自动化,可以以越来越高的通量生成高质量的细胞内冷冻 ET 数据。随后的结构分析的先决条件是可靠地识别一组相对同质的大分子复合物。然而,由于细胞内环境的复杂性和拥挤性,以及 cryo-ET 图像采集带来的限制,例如低信噪比 (SNR) 和不完整的角度采样;因此,这种 3D cryo-ET 体积的数据挖掘仍然是一个主要瓶颈。
cryo-ET 数据集中用于细胞结构分割和大分子复合物(或者说,粒子)定位的一系列可用半自动化方法大致分为基于模板和无模板的方法。模板匹配是一种常用的计算方法,它基于对所讨论复合体的已知模板的相似性系数(互相关)的逐点数值计算。它在大型结构的定位方面是准确的,但无法识别较小或密度较低的粒子,并且计算量大。当前使用经典图像处理的无模板方法专为特定分子结构而设计,因此仅限于特定分子结构,其中颗粒与大细胞结构(如膜或微管)相关联。这两种方法通常都需要人工检查,因此既费力又费时。
深度学习方法,特别是卷积神经网络(CNN)的出现,为 cryo-ET 中的分割和粒子定位提供了更通用和自动化的方法。第一种方法是二维(2D)CNN,它对核糖体或膜等大型结构进行语义分割。剩余的限制包括不太普遍的粒子的定位,以及在其细胞环境中对获得的预测的解释。
在这里,德国海德堡 EMBL 的研究人员介绍了 DeePiCt,这是一种开源软件,可协同监督卷积网络以分割细胞区室(细胞器或胞质基质)和结构(膜或细胞骨架)以及粒子定位。该团队生成了一组全面的专家注释断层扫描图,这些断层扫描图是在野生型裂殖酵母的低温聚焦离子束(cryo-FIB)薄片上采集的,用于新方法的训练和基准测试。


图示:DeePiCt 2D 和 3D CNN 架构在自动化工作流程中实施,结合了低温 ET 数据中的隔室分割和粒子定位。(来源:论文)
DeePiCt 工作流程有助于准确快速地定位完整细胞的低温 ET 数据中的不同结构。3D CNN 架构的高性能和灵活性为模式识别提供了可靠的工具。这使研究人员能够检测到与核糖体相比结构特征(FAS)密度较低的低丰度粒子种类。结构分割(由 3D CNN 预测)与上下文信息(由 2D CNN 预测)的集成排除了粒子定位和结构分割任务中的误报,并利用细胞环境开展以生物兴趣区域为重点的空间研究。这使研究人员能够研究靠近特定细胞器的核糖体(例如,ER 与线粒体)并获得具有功能意义的结构见解。由于代码是开源的并且基于 Python,这里灵活的 3D 架构可以通过实施变体进一步扩展,例如 ResNet 编码器、空洞卷积、类归一化、将 DeePiCt 定位为深度学习技术的测试器。


图示:非合成数据的基准真相的迭代综合注释。(来源:论文)
监督机器学习领域的一个主要瓶颈是专家策划的训练数据的可用性。该团队提供了在两种显微镜采集设置(VPP 和散焦)下的 20 个 S. pombe 断层照片的实验性 cryo-ET 数据集,以及核糖体和 FAS、膜、细胞器和胞质溶胶分割的高质量综合注释。这构成了该领域中第一个足够大的模型训练的黄金标准数据集,这将能够对当前方法进行基准测试,并促进未来计算工具的发展,以在 cryo-ET 数据中进行无偏数据挖掘。来自基准真相或 DeePiCt 预测的注释粒子的子断层图平均产生了 S. pombe 核糖体和脂肪酸合酶的第一密度图,并进一步指出 VPP 或散焦断层扫描的子断层分析的差异,尽管两者都来自野生型 S. pombe cryo-FIB 薄片。
研究人员对 DeePiCt 性能的分析证实,数据质量对其预测能力很重要。这是专门针对核孔复合物(NPC)的预测而证明的,NPC 在细胞内的亚基和孔径水平上具有高度的结构灵活性,是一个具有挑战性的目标。高 SNR 和对比度对于使用 DeePiCt 进行训练和预测期间的良好性能总体而言非常重要,例如使用 VPP 获取的数据具有更高的性能。预处理均衡滤波器的引入改进了 3D 分割网络训练期间的学习过程,用于打印密度低于核糖体的粒子(例如,FAS),特别是跨域的泛化能力,包括不同的显微镜采集条件。

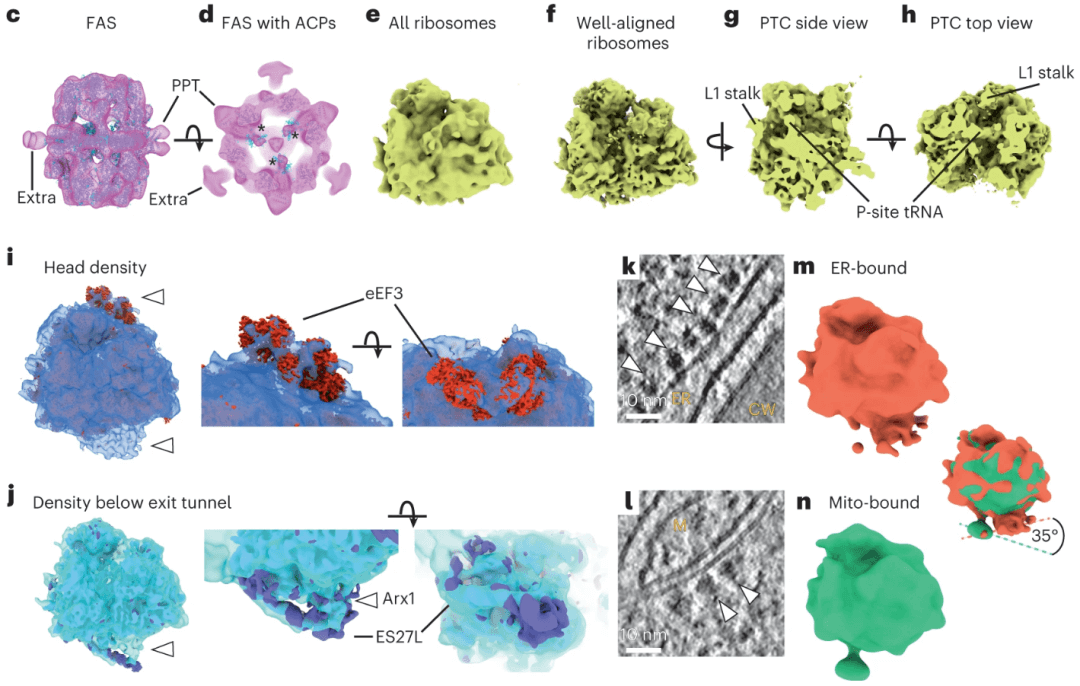
图示:DeePiCt 能够在其细胞环境中探索大分子复合物。(来源:论文)
对于2D网络,尽管预处理没有提高同一域推断的模型性能,但当对VPP数据进行训练和对散焦数据进行推断时,或者当对两种数据类型进行训练以分割细胞器和胞浆时,预处理确实提高了跨域性能。更复杂的任务,例如预测单个细胞器类型,可能需要更多的训练数据或训练具有定制网络架构的专用 3D CNN。
这里的工作流程可以轻松适应其他结构的分割,如细胞骨架分割网络的应用所示。在使用细丝的宽方向采样数据进行训练后,网络在 HeLa 细胞数据集中显示出高质量的微管性能,产生可用于后续结构分析的分割。肌动蛋白预测显示 F1 分数较低,因此可能需要更多的训练数据,并对结构特征的不同方向进行采样,以提高性能。
总而言之,将多个分割网络应用于 HeLa 细胞数据集表明,在来自不同显微镜、物种和条件的数据集上训练的 DeePiCt 模型在高质量数据中产生了相当好的结果。尽管需要进行更深入的分析来研究DeePiCt网络在其他数据集上的适用性限制,但论文里的结果构成了使用冷冻ET对来自不同实验室和公开可用数据集的细胞进行结构生物学大规模定量分析的第一步。

图示:DeePiCt 跨物种泛化。(来源:论文)
从这个意义上说,该研究中生成的基准真相注释,为社区提供了改进和进一步开发 cryo-ET 对象分割和检测工具的资源,最终能够在其细胞环境中广泛探索粒子。结合训练有素的网络和 DeePiCt 工作流程的灵活性,该软件在未来定量冷冻 ET 研究方面具有巨大潜力。
论文链接:https://www.nature.com/articles/s41592-022-01746-2
相关报道:https://phys.org/news/2023-02-ai-scientists-decipher-cellular.html