Nature
公众号/ ScienceAI(ID:Philosophyai)
编辑 | 萝卜皮

当前,迫切需要发现新结构类别的抗生素来解决持续存在的抗生素耐药性危机。深度学习方法有助于探索化学空间;这些通常使用黑盒模型并且不提供化学见解。
麻省理工学院(MIT)的研究人员开发了一种用于抗生素发现的 深度学习 方法,并表明它可以从大型化学库中识别出潜在的抗生素。研究人员用该方法从药物再利用中心(包含约 6,000 个分子)中发现了 halicin 和 abaucin,并从 ZINC15 库中的约 1.07 亿个分子中发现了新的抗菌化合物。

图示:Yann LeCun 转发了这项研究的 Twitter 报道。(来源:网络)
这种方法依赖于 Chemprop,这是一个图 神经网络 平台,通常是黑盒模型,或者不容易解释或解释的模型。根据定义,解释此类模型揭示了模型为得出预测而执行的决策步骤的模式(interpretability),或使此类预测可供人类理解(explainability)。
「这里的见解是,我们可以看到模型正在学习什么,以做出某些分子将成为良好抗生素的预测。从化学结构的角度来看,我们的工作提供了一个具有时间效率、资源效率和机械洞察力的框架,这是我们迄今为止所没有的。」麻省理工学院医学工程与科学研究所(IMES)和生物工程系医学工程与科学 Termeer 教授 James Collins 说道。
IMES、哈佛大学博德研究所的博士后 Felix Wong 和 Collins 指导下的前哈佛医学院研究生 Erica Cheng 是该研究的主要作者,该研究是 MIT Antibiotics- AI 项目的一部分。
该研究以「Discovery of a structural class of antibiotics with explainable deep learning」为题,于 2023 年 12 月 20 日 发布在《Nature》。

迫切需要发现新的抗生素
持续的抗生素耐药性危机可能导致现有抗生素失效,并增加细菌感染的发病率。由于缺乏新的抗生素,这场危机变得更加严重,如果没有新的抗生素,预计到 2050 年全球每年因耐药感染而死亡的人数将达到 1000 万人。
与这种急需的状态相反,抗生素的开发一直缓慢且漫长;从 1962 年推出氟喹诺酮类抗生素到 2000 年推出下一个新结构类别恶唑烷酮,中间间隔了 38 年。
在过去的十年中,通过基于天然产物挖掘、高通量筛选、进化和系统发育分析、结构引导和合理设计以及使用机器学习的计算机筛选的各种方法发现了候选抗生素。然而,开发有效的抗生素发现方法,更好地利用化学空间的巨大结构多样性仍然是一个挑战,迫切需要新的抗生素发现方法。
高效的DL模型发现新抗生素
麻省理工学院的研究人员通过开发了一种可解释的、基于化学子结构的方法用于高效、深度学习引导的化学空间探索,从而预测和发现新的抗生素。
该团队确定了 39,312 种化合物的抗生素活性和人体细胞毒性特征,并应用图神经网络集成来预测 12,076,365 种化合物的抗生素活性和细胞毒性。使用可解释的图形算法,该团队确定了预测具有高抗生素活性和低细胞毒性化合物的基于子结构的基本原理。
「你基本上可以将任何分子表示为化学结构,并且你还可以告诉模型该化学结构是否抗菌。」Wong 说,「该模型接受了许多这样的例子的训练。如果你给它任何新的分子、原子和键的新排列,它可以告诉你该化合物被预测具有抗菌性的概率。」
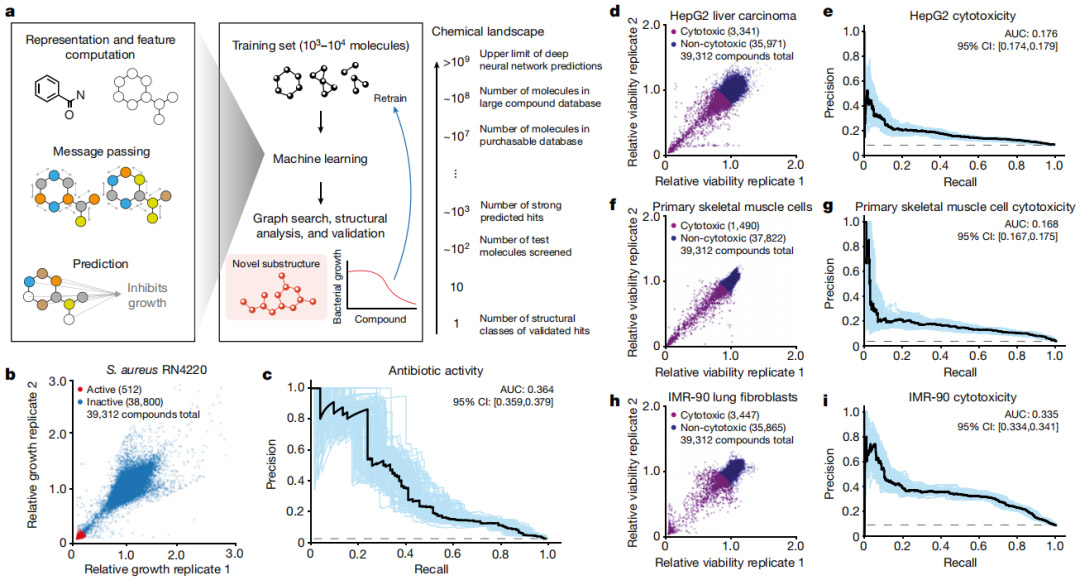
图示:用于预测抗生素活性和人体细胞毒性的深度学习模型集合。(来源:论文)
研究人员凭经验测试了 283 种化合物,发现对金黄色葡萄球菌表现出抗生素活性的化合物在基于基本原理的推定结构类别中得到了丰富。在这些结构类别的化合物中,其中一种对耐甲氧西林金黄色葡萄球菌(MRSA)和耐万古霉素肠球菌具有选择性,可避免实质性耐药性,并降低 MRSA 皮肤和全身大腿感染小鼠模型中的细菌滴度。
「我们有相当有力的证据表明,这种新的结构类别通过选择性地消散细菌中的质子动力,对革兰氏阳性病原体具有活性。」Wong 说,「这些分子选择性地攻击细菌细胞膜,不会对人体细胞膜造成实质性损害。我们大幅增强的深度学习方法,使我们能够预测这种新的抗生素结构类别,并发现它对人体细胞没有毒性。」
对于机理和构效关系的分析表明,该结构类别还可以进一步优化,从而提高对革兰氏阳性病原体的选择性,并增加对革兰氏阴性病原体的渗透性。
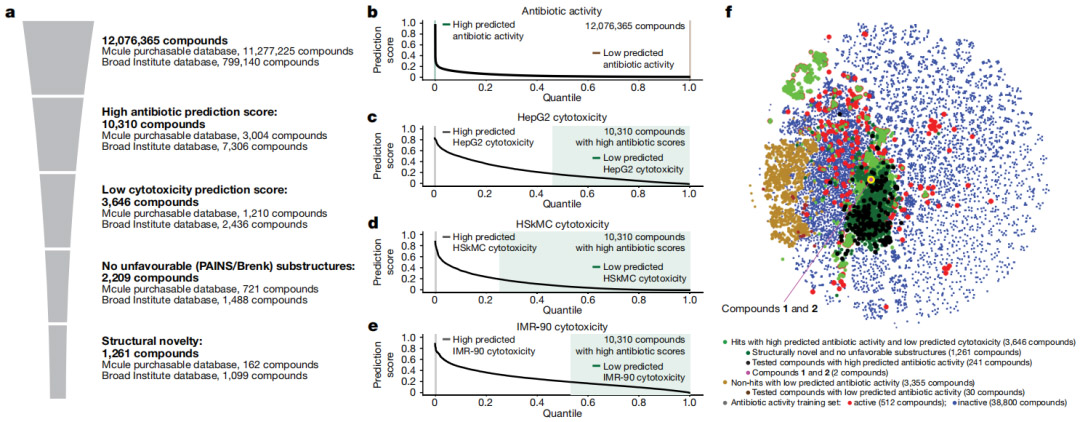
图示:过滤和可视化化学空间。(来源:论文)
作为一种发现抗生素结构类别的深度学习方法,该方法系统地建立在对单独化合物命中预测的基础上,并能够对广阔的化学空间进行有效的、基于子结构的探索。
除了对化学空间进行下采样之外,该方法的一个重要特征是能够自动识别前所未有的结构图案,特别是在深度学习模型的背景下。这种能力提供了化学新颖性的来源,可以建议化学空间来探索和有效地增强当前的发现管线,例如,通过为从头设计工作生成感兴趣的化学片段。
重要的是,这种能力无法使用替代方法来实现,例如传统的 QSAR 分析,这些方法建立在已知的支架上,并且不能基于概括化学结构中分子原子和键的模式来识别新的支架。
研究人员估计,更好地理解基于图表的基本原理预测可以帮助发现和设计其他急需的抗生素类别(例如,那些对革兰氏阴性细菌有活性的)以及针对其他生物过程和疾病的药物类别,包括抗病毒和抗癌药物。
药物发现DL模型变得可解释
该研究的另一个重要意义是药物发现中的深度学习模型可以得到解释。事实上,机器学习中常用的黑盒模型的一个基本限制是,此类模型通常不向底层决策过程提供信息。然而,模型的可解释性可能会带来普遍的见解,从而更好地为探索化学空间的下一代方法的使用和开发提供信息。
该研究表明,使用基于图的搜索来概括模型预测的化学子结构基本原理,可以更好地理解和解释图神经网络。这为特定模型或模型集合所学到的内容提供了有意义的化学见解。
研究人员表示,他们未来的工作将基于这种方法和类似的方法来进一步分析和理解深度学习模型生成的预测,例如,通过使用以扰动模型输入为中心的方法来进行额外的可解释性测试,以及扰动神经网络结构以实现可解释性。
「我们已经在利用基于化学子结构的类似方法来从头设计化合物,当然,我们可以很容易地采用这种开箱即用的方法来发现针对不同病原体的新型抗生素。」Wong 说。
论文链接:https://www.nature.com/articles/s41586-023-06887-8
相关报道:
https://phys.org/news/2023-12-ai-class-antibiotic-candidates-drug-resistant.html
https://www.nature.com/articles/d41586-023-03668-1
https://twitter.com/Nature/status/1737693089056129456