公众号:
编辑 | 萝卜皮
偶极扩散函数 (DSF) 工程重塑了显微镜的图像,可以最大限度地提高测量偶极状发射器 3D 方向的灵敏度。然而,严重的泊松散粒噪声、重叠图像以及同时拟合高维信息(包括方向和位置)使单分子定向定位显微镜(SMOLM)中的图像分析变得非常复杂。
在这里,华盛顿大学的研究人员报告了一种基于深度学习的估计器,称为 Deep-SMOLM,它在理论极限的 3% 内实现了卓越的 3D 方向和 2D 位置测量精度(3.8° 方向、0.32 sr 摆动角和 8.5 nm 横向位置,使用 1000 个检测到的光子)。
Deep-SMOLM 还展示了对发射器重叠图像的最新估计性能,例如,发射器相隔 139 nm 的 Jaccard 指数为 0.95,对应于 43% 的图像重叠。Deep-SMOLM 从包含高度重叠的 DSF 的图像中以比迭代估计器快约 10 倍的速度准确地重建模拟生物纤维和实验淀粉样蛋白原纤维的 5D 信息。
该研究以「Deep-SMOLM: deep learning resolves the 3D orientations and 2D positions of overlapping single molecules with optimal nanoscale resolution」为题,于 2022 年 9 月 26 日发布在《Optics Express》。
单分子定向定位显微镜(SMOLM)是一种通用工具,用于可视化生物分子之间的相互作用及其所产生结构的结构;它以纳米级分辨率同时测量单个荧光分子的 3D 方向和位置。研究人员已经使用分子取向来阐明淀粉样蛋白聚集体的结构、肌动蛋白丝中蛋白质的组织,以及胆固醇引起的脂质膜极性和流动性的变化。
为了在单分子(SM)成像中有效使用有限的光子预算,必须设计偶极子扩展函数(DSF),即光学显微镜点扩展函数的矢量扩展,以编码有关分子 3D 方向的附加信息。然而,同时估计发射器的 3D 方向和位置具有挑战性,因为(1)很难在 5 维空间(3D 方向、摆动、2D 位置)中估计 SM 参数而不陷入由严重的泊松散粒噪声引发的局部最小值;(2)工程 DSF 具有较大的足迹,导致 SM 图像频繁重叠;以及(3)当大型 DSF 将光子散布在许多相机像素上时,暗发射器很难检测到。
为了估计 SM 方向,现有技术将嘈杂的实验图像与预先计算的采样 DSF 库进行匹配,或者使用成像系统的模型构建参数拟合。这些方法要么遭受(1) 由于有限采样和/或 DSF 近似而导致精度降低或(2) 在缓慢的迭代优化过程中计算负担过重。
此外,估计图像重叠的暗淡 SM 的参数极具挑战性。用于测量方向的早期神经网络仅限于包含一个发射器的图像。最近,DeepSTORM3D 和 DECODE 已被开发用于估计单个分子的 3D 位置,即使对于图像重叠的高密度发射器也是如此。然而,仍然缺少能够从密集发射器的重叠图像进行高维估计的技术,即测量五个或更多参数。
在这里,华盛顿大学的研究人员展示了一种基于深度学习的估计器,称为 Deep-SMOLM,用于从实现工程化偶极子扩展功能的显微镜中同时估计单个分子的 3D 方向和 2D 位置。与传统的优化方法相比,Deep-SMOLM 对 3D 方向和 2D 位置均实现了卓越的估计精度,平均在最佳精度的 3 以内(图 2(b-d))。通常,为高维估计设计损失函数并特别是在多个参数之间平衡权重总是具有挑战性的。
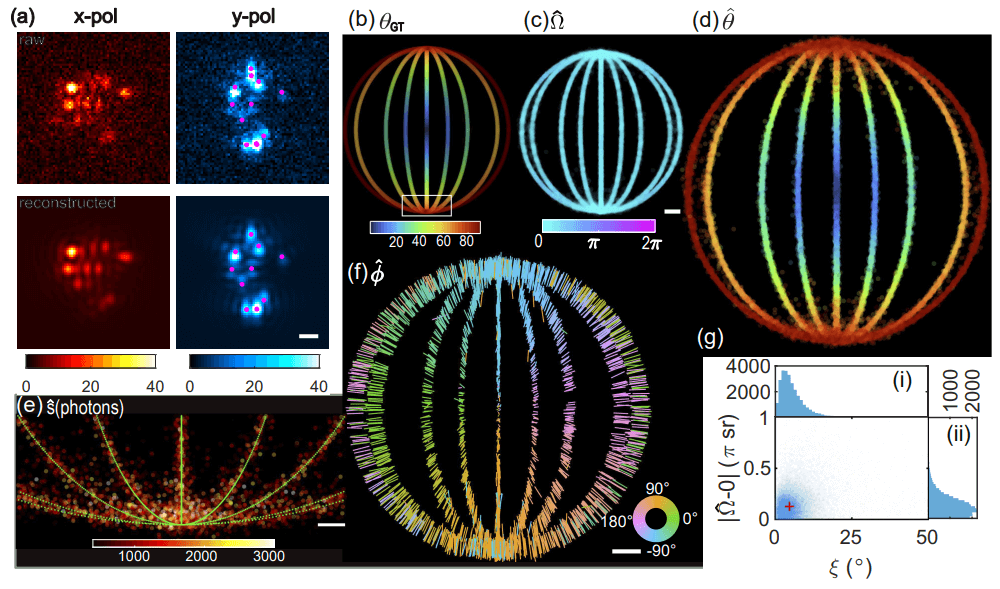
图示:使用 Deep-SMOLM 估计单分子 (SM) 的 3D 方向和 2D 位置。(来源:论文)
研究人员将 Deep-SMOLM 的卓越性能归因于从嘈杂的 SM 图像中估计亮度加权定向二阶矩的线性;否则,直接估计方向角 %[,,Ω] 是病态且不稳定的。重要的是,对于高性能 DSF,每个亮度加权方向矩对最终 DSF 形状的贡献大致相同。
图示:Deep-SMOLM 用于估计 SM 的 3D 方向和 2D 位置的精度。(来源:论文)
另外,研究人员设计了 3D 方向和 2D 位置,分别正交编码为 Deep-SMOLM 输出图像中高斯点的强度和空间位置。这些设计策略使得在训练 Deep-SMOLM 时不需要调整六个输出图像之间的权重,并且在 5 维估计之间得到的训练非常平衡。
图示:1D 纤维模型结构的 Deep-SMOLM 5D 成像。(来源:论文)
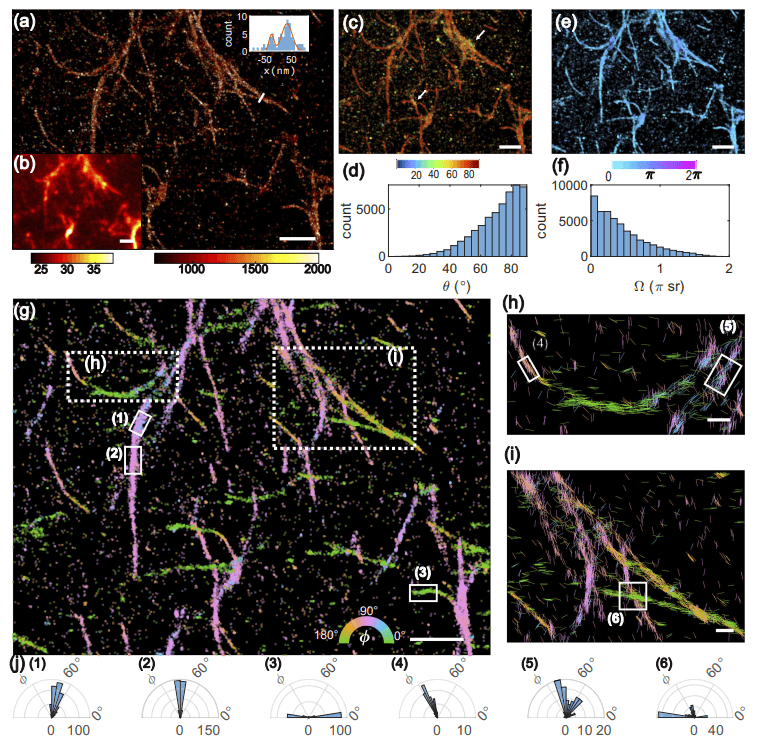
正如模拟结构和实验性淀粉样蛋白原纤维所证明的,Deep-SMOLM 在估计重叠 DSF 方面表现出出色的性能,例如,密度约为 0.6 个发射器/µm^2,比传统的基于优化的算法所允许的密度高约 2 倍。这种能力应该允许 Deep-SMOLM 通过使荧光探针以更高的速率闪烁并以更高的浓度使用,从而在 SMOLM 数据采集中实现约 2 倍的加速。
图示:与 Aβ42 淀粉样蛋白纤维瞬时结合的尼罗红 (NR) 的 5D SMOLM 图像。(来源:论文)
另外,Deep-SMOLM 需要相对较少的训练时间和数据(约 2 小时和 30,000 张噪声图像,包含约 330,000 个总发射器)。经过训练,Deep-SMOLM 在每帧基础上估计 3D 方向和 2D 位置的速度比 RoSE-O 等迭代算法快约 10 倍(Deep-SMOLM 约 30 秒,RoSE-O 分析 1,000 帧约 6 分钟) 。考虑到计算可靠 SMOLM 重建所需的原始帧更少,与迭代算法相比,Deep-SMOLM 的计算时间总体上提高了 20 倍,以获得相同的定位总数。
研究人员注意到帧之间 SM 闪烁的相关性也可用于进一步增强 Deep-SMOLM 在估计重叠发射器方面的性能。他们预计 Deep-SMOLM 将使动态过程的快速高维 SMOLM 成像成为可能,并可能在亚分钟时间尺度上发现结构变化。
在不久的将来,该团队计划扩展 Deep-SMOLM 以同时估计重叠分子的 3D 位置和 3D 方向;由于 pixOL 的基础图像对每个发射器的轴向位置具有很强的敏感性,这种分析本质上比 2D SMOLM 复杂得多。
另外,尤其是在 3D 中对体内细胞结构进行成像时,估计器需要提高对模型不匹配的鲁棒性,例如,适应非均匀背景以及场和深度相关的光学像差。然而,详尽地训练网络以预测成像系统的所有实际扰动是极具挑战性的。因此,同时学习模型不匹配以及估计 3D 位置和 3D 方向可以提高 Deep-SMOLM 在具有挑战性的成像条件下的性能。这种自适应方法可能是确保网络对于体内超分辨率成像具有足够泛化性的关键,并且可以释放 SMOLM 在细胞和组织尺度成像方面的全部潜力。
论文链接:https://opg.optica.org/oe/fulltext.cfm?uri=oe-30-20-36761&id=505938
相关报道:https://techxplore.com/news/2022-11-machine-pictures-proteins-5d.html